Fechamento – 12:00 – 25/10/01
25 de outubro de 2001Liberação das exportações de carne do RS para UE pode estar próxima
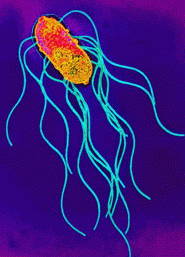
26 de outubro de 2001Genoma de Salmonella é seqüenciado
Duas equipes de cientistas seqüenciaram o genoma de duas cepas da bactéria Salmonella – uma delas é responsável pela febre tifóide; a outra causa intoxicação alimentar. A identificação do genoma poderá auxiliar no diagnóstico, tratamento e vacinação contras ambas as doenças. Além disso, a comparação dos genomas permitirá detectar porque duas bactérias aparentemente tão parecidas provocam doenças tão diferentes nos seres humanos.
As duas cepas são chamadas de typhi e typhimurium. A typhi, bactéria causadora da febre tifóide, afeta somente seres humanos, acometendo o fígado, o baço e a medula óssea. Já a thyphimurium, que acomete principalmente os intestinos, é a principal causa de intoxicação alimentar provocada por Salmonella, apresentando sintomas menos graves. Segundo o microbiologista da Universidade de Illinois, Stanley Maloy, essa bactéria acomete praticamente todos os seres vivos.
A febre tifóide afeta cerca de 16 milhões de pessoas por ano, com cerca de 600 mil casos de morte. Além disso, sua resistência à drogas está tornando-se cada vez mais preocupante. A equipe que seqüenciou a Salmonella typhi utilizou uma cepa do Vietnã, que é resistente a vários antibióticos.
Os dados presentes no genoma seqüenciado poderão aumentar as ferramentas de diagnóstico da doença, segundo um membro da equipe de pesquisa, Gordon Dougan, do Imperial College, em Londres. A febre tifóide é difícil de ser diagnosticada porque seus sintomas podem ser confundidos com os de outras enfermidades, incluindo a malária e a dengue. Além disso, a bactéria é difícil de ser reconhecida.
A vacina contra a febre tifóide não é totalmente confiável, e não está incluída nos programas de vacinação infantil. “Nós precisamos avançar mais na vacinação”, disse Dougan. Caso isso ocorra, a doença poderá ser totalmente erradicada, segundo informou o líder do projeto genoma da Salmonella typhi, Julian Parkhill, do Sanger Centre em Cambridge, Reino Unido.
A Salmonella typhi tem mais de 200 “pseudogenes” – pedaços de DNA que foram inativados pela mutação. As versões funcionais desses genes foram inativadas durante a evolução e o estabelecimento deste microrganismo em seu habitat atual.
Já a bactéria Salmonella typhimurium, tem apenas 40 pseudogenes. Cada uma das bactérias tem centenas de genes que não são encontrados na outra. “Para dois organismos que são classificados como da mesma espécie, a quantidade de diferenças que existe entre eles é surpreendente”, disse Parkhill.
Problemas e soluções
A S. thyphimurium tem uma imagem menos alarmante junto ao público, mas é um problema maior para a saúde pública do que a S.typhi, disse o líder da pesquisa que seqüenciou a S. typhimurium, Michael McClelland, do Sydney Kimmel Cancer Center, em San Diego, Califórnia. “Provavelmente, há centenas de milhares de casos de intoxicação por esta bactéria todos os anos no mundo.” Além disso, ele acrescentou que uma intoxicação alimentar provocada por esta bactéria pode matar 2 vezes mais – principalmente crianças e idosos – do que a febre tifóide.
O seqüenciamento da S. thyphimurium revelou 50 genes anteriormente desconhecidos, que codificam a produção de proteínas de superfície. Esses são alvos potenciais para o desenvolvimento de vacinas e drogas.
A bactéria, ao mesmo tempo que é uma ferramenta de estudo, é uma ameaça. Versões enfraquecidas do microrganismo são usadas para desenvolver vacinas e drogas. Em ratos, os sintomas causados pela S. thyphimurium são muito parecidos com a febre tifóide humana. A equipe que seqüenciou o genoma desta bactéria pretende colocar nelas microchips para identificar os genes que fazem com que a bactéria aja de forma diferente em diversas situações. Isso poderá explicar, por exemplo, porque ela é capaz de acometer quase todos os seres vivos, e também, porque seus efeitos são diferentes no homem e no rato.
Existem mais de 2 mil cepas de Salmonella que agem no intestino e que acometem vários animais, bem como o homem. O genoma da maioria das cepas está sendo seqüenciado, e a comparação entre todas elas poderá ser bastante útil futuramente.
Referências
1. Parkhill, J. et al. Complete genome sequence of a multiple drug resistant Salmonella enterica serovar Typhi CT18. Nature, 413, 848 – 852, (2001).
2. McClelland, M. et al. The complete genome sequence of Salmonella enterica serovar Typhimurium LT2. Nature, 413, 852 – 856, (2001).
Fonte: Nature News Service, adaptado por Equipe BeefPoint


